nap パッケージで用いるpythonモジュール群.
どのpythonスクリプトも大抵 -h オプションをつけて実行すればヘルプが表示される.
napsys.py
ファイル形式の変換(convert)や原子構造ファイルの解析(analyze)を行う.
-hオプションをつけると使い方が表示される.
convert
$ python /path/to/nappy/napsys.py convert --specorder=O,Li,Zr,P POSCAR pmdini上記の例では,POSCARファイルをpmd形式の「pmdini」という名前のファイルに変換している.
「-h」オプションを付けて実行するとヘルプが表示される. 現在対応しているフォーマットは,以下.
- pmd
- POSCAR
- dump
- xsf
コマンドへのパスを省略したい
毎回
/path/to/nappy/napsys.pyをタイプするのが面倒な人は,環境変数PATHに~/bin/を登録しておいて,スクリプトへのリンクを貼る.$ cd ~/bin $ ln -s /path/to/nappy/napsys.py .こうすることでシステム内のどこからでも
napsys.pyとタイプするだけで実行できるようになる(Linux/Unix/macOS).
系のサイズを変更する
「
--periodic copy=2,2,2」というオプションをつけると,入力された系を 2x2x2 に拡張する.
analyze
$ napsys.py analyze --specorder=Li,Zr,P,O POSCAR
a1 vector = [ 7.744, 0.000, -4.256]
a2 vector = [ -4.994, 7.353, -0.181]
a3 vector = [ 0.000, 0.000, 9.161]
a = 8.837 A
b = 8.890 A
c = 9.161 A
alpha = 91.17 deg.
beta = 118.79 deg.
gamma = 118.85 deg.
volume= 521.632 A^3
number of atoms = 36
number of atoms per species:
Li: 2
Zr: 4
P: 6
O: 24
density = 3.02 g/cm^3このようにセル情報を表示してくれる.
POSCARにすでに元素情報が入っていても,現状では --specorder=Li,Zr,P,O のように元素を指定しなければならない.
vasp/prepare.py
POSCARファイルを指定して,このスクリプトを実行すると,VASP計算に必要な以下のファイルを作成してくれる.
- INCAR
- KPOINTS
- POTCAR
-h をつけて実行するとヘルプが表示され,VASP計算に関するいくつかの指定ができることが分かる.VASP計算に関するメモは http://ryokbys.web.nitech.ac.jp/vasp.html などを参照のこと.
POTCARは,VASPが提供しているPOTCARのセットをどこかに置いておき,そこを参照するように指定する.デフォルトでは,~/local/vasp/potpaw_PBE というディレクトリに各種元素の擬ポテンシャル情報が置かれているとしている.--potcar-dir オプションで指定することもできる.
vasp/vasprun2fp.py
VASP計算の結果から,各MDステップもしくは各構造緩和ステップにおけるエネルギー,力,応力などを抽出する.-h でオプションを確認できる.
MDもしくは構造緩和をした(C,O,Hの)系の場合,
$ python /path/to/nap/nappy/vasp/vasprun2fp.py --specorder=C,O,H --sequence ./このようにして,指定したディレクトリ(./)にあるvasprun.xmlから,各ステップのデータを抽出し,00001,00002…というディレクトリに格納する.
Jupyter notebook で利用する
nappy パッケージがインストールされていることを前提としている.インストールは,nap doc に書かれているように,napのルートディレクトリ(pmdやnappyのディレクトリがあるところ)で,下記コマンド実行する.
$ python setup.py sdist
$ pip install -e .モジュールのロード
下記のようにして nappy パッケージを読み込む.(実際には nappy.napsys.NAPSystem クラスを利用する)
import nappyファイルの読み書き
nsys = nappy.io.read('POSCAR')
nappy.io.write(nsys,'POSCAR_new')- バージョンが古いと上のやり方ができないのでエラーが出ると思われる.
- ファイルの読み込みと同時に
NAPSystemオブジェクトを作成し,nsysに代入する. nappy.io.write()関数はファイル名から形式を推測してファイルに書き出す.
NAPSystem オブジェクト
nsys という名前に代入された NAPSystem オブジェクトは次のデータを持っている.
- alc : 格子ベクトルに掛ける因子
- a1,a2,a3 : 格子ベクトル(これに alc を掛けると実格子ベクトル).numpy array オブジェクト.
- specorder : この系における元素の順番
- atoms : 原子情報(pandas.DataFrameオブジェクト)
dir(nsys)とすることで,nsys オブジェクトがどのような変数や関数を持っているか見ることができる.
以下のいくつかの例でこれらを説明する.
analyze information
上で説明したanalyze情報を表示することができる.
print(nsys)specorder
print(nsys.specorder)
...
['C','O','H']上のように元素名のリストとなっている.
atoms
Jupyter notebookで,
nsys.atomsとすると,下図のようなpandas.DataFrameの中身が出力される.
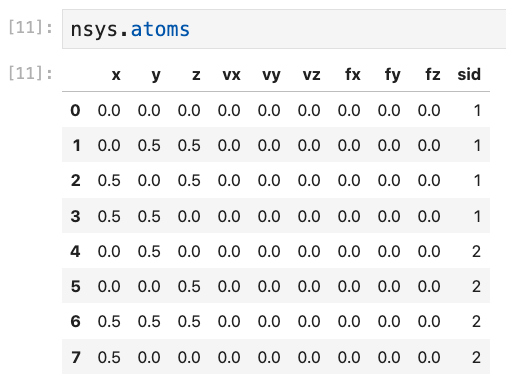
この各行が各原子に対応しており,インデックスは0から始まっている. 各列の情報は以下:
- x,y,z : 各原子の座標
- vx,vy,vz:速度.読み込んだファイルに情報がなければ0.
- fx,fy,fz:力.読み込んだファイルに情報がなければ0.
- sid:原子の species ID.nsys.specorder に対応(1,2,3,…).sidの「1」は,specorderの0番に対応することに注意.つまり,
nsys.specorder[sid-1]とすると,対応する元素名が取得できる.
add_atoms
原子(群)を追加する.次のように,元素情報と座標情報を指定して追加する.
symbols = ['Li', 'F']
poss = [[0.5, 0.6, 0.7],
[0.5, 0.6, 0.9]]
nsys.add_atoms(symbols, poss)速度や力なども同時に指定したければオプション指定可能.
pandas.DataFrame の使い方の例
8番原子のx座標や(x,y,z)座標を取得する.
x = nsys.atoms.at[8,'x']
# もしくは
x = nsys.atoms.loc[8, ['x','y','z']]ただし,原子インデックスは 0 から始まることに注意.
元素番号2の原子データを抽出する
nsys.atoms[nsys.atoms.sid == 2]
# もしくは,
nsys.atoms.query('sid == 2')特定原子の座標を変更する
# 10番原子の座標を変更する.
pos = nsys.atoms.loc[10,['x','y','z']] = [0.1, 0.5, 0.0]
# ちゃんと変更が反映されていることを確認
nsys.atomsこれをファイルに出力すれば,原子座標の値を変更して,原子を操作するpythonスクリプトが完成する.
atom.py
以下のように,元素名を指定して実行すると,それらの元素に関するいくつかの情報を表示する.
$ python /path/to/nappy/atom.py O Li Zr P
Element: O
name: Oxygen
atomic number: 8
mass: 15.999
abundance: 461000
Element: Li
name: Lithium
atomic number: 3
mass: 6.940
abundance: 20
Element: Zr
name: Zirconium
atomic number: 40
mass: 91.224
abundance: 165
Element: P
name: Phosphorus
atomic number: 15
mass: 30.974
abundance: 1050mendeleev というパッケージを使うとより豊富な情報を表示してくれる.参考までに.